Zusatzinformationen
Trennung von Nukleinsäuren | Ethidiumbromid | Nukleinsäurequantifizierung | alternative DNA-Farbstoffe
Trennung von Nukleinsäuren
Es ist grundsätzlich zu unterscheiden, ob z.B. Oligonukleotide, Plasmide, Megaplasmide oder sonstige möglichst langen DNA-Fragmente aufgetrennt werden sollen. Eine Übersicht zu den anwendbaren Verfahren gibt Tabelle 1.
| Tabelle 1: Trennung von Nukleinsäuren | ||
| Medium | %-Zusammensetzung | Trennbereich (Bp) |
| Agarose | 0,3 | 50 000 - 1000 |
| 0,7 | 20 000 - 300 | |
| 1,4 | 6000 - 200 | |
| Acrylamid | 4 | 1000 - 100 |
| 10 | 500 - 25 | |
| 20 | 50 - 1 | |
Für sehr kurze Fragmente werden also in der Regel Polyacrylamidgele als Trennmedium benutzt. Diese Acrylamidgele haben den Vorteil gegenüber Agarosegelen, dass auf ihnen auch eine ungefährliche, empfindliche Silberfärbung durchgeführt werden kann. Im Agarosegel würden die reduzierenden Zuckergruppen bei der Silberfärbung zu einem starken Hintergrund führen, auf dem die gefärbten DNA-Banden kaum erkennbar wären. Acrylamidgele werden auch zum Auftrennen von Produkten der Nukleotidsequenzierreaktionen benutzt. Bei Sequenzierungen steht im Vordergrund, Fragmentlängen auftrennen zu können, die sich um ein Basenpaar unterscheiden.
Ethidiumbromid
DNA-Banden in Agarosegelen werden häufig durch Ethidiumbromid gefärbt. Die nachfolgende Abbildung zeigt die Strukturformel dieser Verbindung, die in Laboren häufig mit "EtBr" abgekürzt wird.
Abbildung 1: Strukturformel des Ethidiumbromides
Dieses Molekül ist eben. Daher kann es zwischen die Basen der DNA-Doppelhelix eingelagert werden. Erst dieser Komplex zeigt durch Anregung mit UV-Licht die typisch orangefarbene Fluoreszenz. Fluoreszenz ist hier, wie in vielen anderen Fällen auch, ein sehr empfindliches Nachweisverfahren.
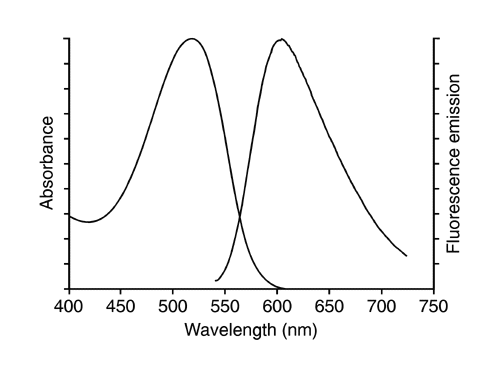
Abbildung 2: Absorptions- und Emissionspektrum von Ethidiumbromid interkaliert in DNA.
Beschreibungen des Ethidiumbromides sind in der folgenden Tabelle zusammengestellt.
| Tabelle: Angaben zu Ethidiumbromid | |
| Summenformel | C21H20BrN3 |
| Molekulargewicht | 394,31 |
| CAS | 1239-45-8 |
| Systematische Bezeichnung | Phenanthridinium, 3,8-diamino-5-ethyl-6-phenylbromide |
Zu Sicherheitsaspekten im Umgang mit Ethidiumbromid kann ein Sicherheitsdatenblatt bzw. ein Merkblatt heruntergeladen werden.
Nukleinsäurequantifizierung durch ein UV-Spektrum. Die O.D.260 = 1 ergibt
- 50µg/ml doppelsträngige (ds) DNA
- 40 µg/ml einzelsträngige (es) DNA
- 33 µg/ml RNA
- Bei Oligos mit bekannter Nukleotidsequenz sollte der Molare Extiktionskoeffizienten der einzelnen Basen bevorzugt für die Quantifizierung genutzt werden.
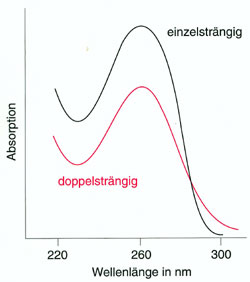
Abbildung 3: Mittleres Absorptionsspektrum der Nukleinsäuren.
Ein solches UV-Spektrum ergibt aus dem Quotienten von E260 /E280 auch einen Hinweis auf Verunreinigungen. Eine erhöhte Absorption bei 280 nm wird durch aromatische Verbindungen wie die Aminosäure Tyrosin (Protein) oder durch Phenol verursacht. Beide Verunreinigungen können den Restriktionsverdau einer solchen DNA stören.
DNA gilt als rein, wenn der Quotient aus E260 /E280 ≥ 1.8 beträgt. Bei 50% DNA und 50% Protein wird E260/E280 ≈ 1.5 gemessen. Für reine RNA beträgt der Quotient aus E260 /E280 etwa 1.95 - 2.15.
Auf solche UV-Messungen wird häufig verzichtet, da die "normalen" Laborphotometer mit ihren Standardküvetten viel DNA in relativ großen Volumina für solche Messungen erfordern würden.
Alternative DNA-Farbstoffe
In der Regel wird doppelsträngige Nukleinsäure gefärbt. Azur B (7-Dimethylamino-3-methylimino-3H-phenothiazion-hydrochlorid, Azur I, CAS 531-55-5, C.I. 52010), der nahe verwandte Farbstoff Methylenblau und SybrGreen bzw. SybrSafe.
SybrGreen und insbesondere SybrSafe (pdf-Datei, 135 KB) gilt als weniger "gesundheitsschädlich" als Ethidiumbromid. Er zeichnet sich sogar durch eine verbesserte Empfindlichkeit aus und die damit gefärbte DNA ist im langwelligen Licht sehr leicht mit einer normalen digitalen Kamera zu dokumentieren. Allerdings gibt es auch entscheidende Nachteile: SybrGreen ist deutlich teurer als Ethidiumbromid, ist photooxidierbar und es gibt von Seiten des amerikanischen Herstellers weder Angaben zur Chemie der Verbindung noch ein Sicherheitsdatenblatt.
