Nachweis einer mehrfachen Antibiotikaresistenz
Ergebnisse
Die Analyse des PCR-Produktes zeigt ein Amplifikat von etwa 500 Bp Länge.
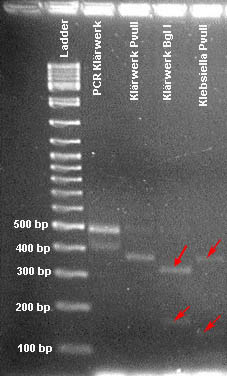
Abbildung 1: 2,5%-iges Agarosegel zur Analyse des PCR-Produktes aus einer Wasserprobe (Auslauf eines Klärwerks). Ladder: Längenstandard in Basenpaaren (bp). Das Gel wurde mit Ethidiumbromid gefärbt.
Die Länge des unverdauten PCR-Produktes entspricht mit 484 Bp der Erwartung, wenn das Gen aus Pseudomonas aeruginosa (ncbi, Zugriffsnummer U12338, Nukleotidsequenz) zu Grunde gelegt wird. Auch die Verdaue mit den Restriktionsendonukleasen PvuII und BglI ergeben die mit Nebcutter vorhergesagten Fragmentmuster. Im Falle von PvuII werden zwei Fragmente der Länge 114 Bp und 371 Bp erwartet; im Falle des Verdaues mit BglI werden Fragmente der Länge 163 Bp und 321 Bp erwartet.
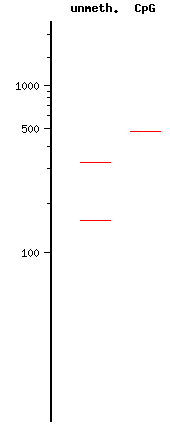
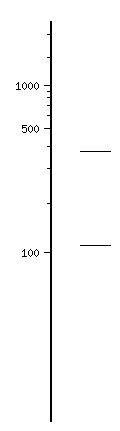
Abbildung 2: Mit dem Computerprogramm Nebcutter vorhergesagte Fragmentierungsmuster des Integrasegens aus Pseudomonas aeruginosa mit den Resitriktionsendonukleasen BglI (links) und PvuII (rechts) in einem 2%-igen Agarosegel.
Die Mischkultur der untersuchten Wasserprobe, aus der die DNA als template für diese PCR verwendet wurde, wurde auf LB-Agarplatten ausgestrichen. Auf diese Agarplatten wurden Testringe mit insgesamt 12 verschiedenen Antibiotika aufgelegt und diese Ansätze über Nacht bei 37°C bebrühtet.


Abbildung 3: Ausstrich der mit PCR untersuchten Mischkultur auf LB-Agarplatten. Auf die Agarplatten wurden zwei verschiedene Antibiotikatestringe mit insgesamt 12 verschiedenen Antibiotika aufgelegt und über Nacht bei 37°C bebrütet.
Es ist zu erkennen, dass die verschiedenen Bakterienarten an die Testplättchen heranwachsen, also nicht durch deren Antibiotikum gehemmt werden. Entstehen Hemmhöfe um die Testplättchen, wie z.B. auf der rechten Agarplatte unten rechts, so finden sich auch auf diesen Flächen Kolonien resistenter Keime.

