Voraussetzungen
Sie sollten mit einer Pipette und mit einer Zentrifuge umgehen können. Falls Sie nicht schon aus einem Züchtungsexperiment eine ausreichende Menge plasmidhaltiger Bakterienzellen besitzen, sollten Sie außerdem Erfahrung mit der Züchtung von E. coli haben, um sich plasmidhaltige Zellen selbst herstellen zu können.
Das Verfahren ist Ihnen zu kompliziert oder Sie haben nicht alle Materialien? Sie möchten dennoch ein Plasmid nachweisen? Dann sollten Sie über das cracking-Verfahren nachdenken.
Zeitbedarf: Für die Isolierung eines Plasmides aus Bakterienzellen benötigen Sie etwa 1 Stunde Zeit. Für die Analyse Ihres Ergebnisses durch die Agarosegelelektrophorese weitere 1,5-2 Stunden. Die Analyse kann unabhängig von der Plasmidisolierung durchgeführt werden.

Folgeexperimente: Eine Agarosegelelektrophorese ist unbedingt zu empfehlen, da Sie nur so den Erfolg ihrer Vector-Präparation überprüfen können. Mit einem Teil der Plasmidpräparation können Sie vor der Agarosegelelektrophorese ausserdem noch einen Restriktionsverdau oder eine Transformation durchführen.

Aufgabe
- Isolieren Sie mit Hilfe eines Reagenziensatzes (kit) das Plasmid pUC18 aus E. coli Zellen.
- Weisen Sie anschließend das Plasmid durch eine Agarosegelelektrophorese nach.
- Vergleichen Sie diese Plasmidpräparation mit der "cracking"-Methode.
Lernziele
Am Beispiel des Sicherheitsvectors pUC18 lernen Sie eine Methode, Plasmide und Vectoren zu isolieren. Sie haben eine Genfähre in Händen, die Sie in E. coli transformieren können, um damit die Eigenschaften dieses Bakteriums zu verändern. Neben den Experimenten des Restriktionsverdaus und der Agarosegelelektrophorese erlernen Sie damit eine wichtige, grundlegende Methode der Klonierungstechnik.
Plasmidisolierung
Zusatzinformationen
Allgemeines | Plasmid pUC18
Allgemeines
Der Vector pUC18 ist ein high copy Plasmid. Es werden bis zu 500 Kopien pro Zelle gebildet. Daher können Sie dieses Plasmid auch besonders leicht nachweisen. Dagegen ist pBR322 ein low copy Plasmid, von dem nur etwa 10-20 Kopien pro Zelle gebildet werden. Low copy Plasmide können wichtig werden, wenn toxische Genprodukte kloniert werden sollen, deren Konzentrationen in der Zelle niedrig gehalten werden müssen.
Vectoren sind "getunte" Plasmide, die im Labor für ihre experimentelle Funktion des Gentransports aus natürlich vorkommenden Plasmiden hergestellt wurden. Um ihre Funktion erfüllen zu können, enthalten Vectoren mindestens drei Gene: ein Reportergen, ein Selektionsgen und den oriV. Als Beispiel ist der pUC18 Vector in der nachfolgenden Abbildung gezeigt, mit dem Sie in diesem Experiment arbeiten und der genau diese Voraussage erfüllt.
Das Plasmid pUC18
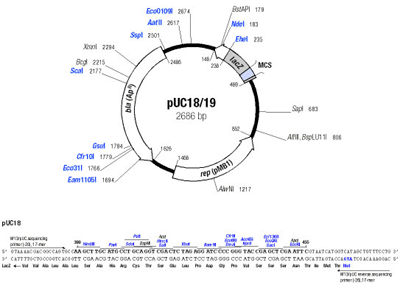
Abbildung 1: Karte des Plasmides pUC18.
Erklärungen zur Vectorkarte: Die dicken Pfeile auf dem Kreis stellen die drei Gene dar. Apr ist das Selektionsgen (bla = beta-Lactamase, Ampicillin-Resistenzgen), lacZ das Reportergen (beta-Galaktosidase) und rep ist der oriV, der die Replikation und den Wirtsbereich des Plasmides bestimmt. MCS ist die multiple cloning site, die unter dem Vector vergrößert wiedergegeben ist. In sie kann Fremd-DNA einkloniert werden. Die anderen Abkürzungen sind Bezeichnungen für Restriktionsendonukleasen, die schwarzen Zahlen dahinter geben die Position der Schnittstelle in der Nukleotidsequenz an.
Ich vermisse noch Zusatzinformationen zu folgenden Stichworten/Gebieten.
Probleme
| Beobachtung | Lösungsansatz |
| Keine Plasmid-DNA im Agarosegel. | Haben Sie Antibiotikum dem Züchtungsmedium zugegeben? Haben Sie einen plasmidhaltigen Stamm verwendet? Nach der DNA-Lagerung: Haben Sie steril gearbeitet oder konnten Nukleasen im Lagergefäß vorhanden sein? |
| Wenig Plasmid-DNA in der Präparation. | Waren alle Zellen bei der Plasmidpräparation resuspendiert? |
| Plasmid-DNA lässt sich nicht mit Restriktionsendonukleasen verdauen. | Haben Sie den Schritt 9 (entfernen des PE Puffers) sorgfältig befolgt? |
Sicherheit
- Sammeln Sie alle Flüssigkeiten und Materialien, die mit plasmidhaltigen Zellen in Berührung gekommen sind, in einem autoklavierbaren Sammelgefäß. Autoklavieren Sie diese Materialien am Ende des Experimentes, bevor Sie Flüssigkeiten im Abguss und die festen Materialien im Hausmüll entsorgen.
- Qiagen empfiehlt zur Handhabe der Puffer P2, N3 und PB Handschuhe zu tragen.
- Vermeiden Sie beim Pipettieren und beim Zentrifugieren die Aerosolbildung.
| Tabelle 1: Sicherheitsdatenblätter als pdf-Dateien | |
| Abkürzungen, Gefahrensymbole, R+S-Sätze | 100 KB |
| Ethidiumbromid | 33 KB |
Tipps und Hiweise
- Zur Züchtung der E. coli Zellen sollten Sie eine Schüttelkultur verwenden. Das Bebrüten in stehenden, nicht belüfteten Kulturen führt zu anaerobem Wachstum. Dabei werden u.a. Säuren gebildet ("gemischte Fermentation"), die zur Erniedrigung des pH-Wertes im Medium führen kann. Beta-Lactamantibiotika wie das Ampicillin werden leicht im sauren Milieu gespalten und damit inaktiviert.
- Achten Sie bei der Zugabe des Ampicillins und des Inokulums darauf, dass die Temperatur des autoklavierten Mediums kleiner als 40°C ist. Sonst kann das Antibiotikum hydrolysiert und die Bakterien während des Inokulierens abgetötet werden.
- Zentrifugieren Sie die ÜN-Kultur nicht zu lange und bei zu hoher Umdrehungsgeschwindigkeit. Der Bakterienniederschlag wird dadurch zu fest. Sie haben dann unnötig Mühe, die Zellen zu resuspendieren (wieder in Lösung zu bringen).
- Nutzen Sie zur visuellen Unterstützung Ihres Experimentes die ausdruckbaren Tischvorlagen und die Powerpoint-Präsentation. Wenn Sie Ihre Tischvorlagen laminieren, dann können die Schüler jeden experimentellen Schritt mit einem Filzschreiber (edding 2000) abzeichnen. Am Ende des Experimentes können diese Anmerkungen mit 80%-igem Ethanol wieder abgewischt werden, die Folie ist erneut verwendbar.
- Verwenden Sie anstelle des Qiagen Kits einen alternativen Reagenziensatz, so müssen Sie den Anweisungen dieses Kit-Lieferanten folgen. Die Prinzipien der Isolierungs-Kits sind zwar alle sehr ähnlich, die einzelnen Schritte unterscheiden sich jedoch im Detail.
- Wenn Sie mehr als 10-20 µg Plasmid-DNA isolieren wollen, so können Sie den Midi (Qiagen 12143) oder gar den Maxi (Qiagen 12162) Kit verwenden. Damit können Sie 100 µg oder bis zu 2000 µg Plasmid-DNA isolieren.
- Das Säulenmaterial des Mini Kits ist modifiziertes Silica.
- Vermeiden Sie Scherkräfte wärend der Plasmidsiolierung. Durch Scherkräfte "nicken" Sie die Plasmide. Scherkräfte entstehen beim kräftigen Mischen (z.B. auf einem Vortexer) oder auch beim Pipettieren.
- Wenn Sie weitere NUGI-Experimente, wie z.B. das GFP- Klonierungsexperiment planen, so können Sie dieses Plasmidisolierungsexperiment auch mit pHAT-GFPuv-haltigen E. coli Zellen durchführen. Dadurch ersparen Sie sich eine erneute Züchtung des pHAT-GFPuv-haltigen E. coli Stammes zu einem späteren Zeitpunkt, um gereinigtes Plasmid für eine Klonierung zu bekommen.
- Wenn Sie das isolierte Plasmid für weitere Experimente lagern möchten, so sollten Sie im letzten Präparationsschritt (Elution des Plasmides von der Säule) nicht Wasser, sondern TE-Puffer (Tris-EDTA-Puffer) verwenden. Das EDTA komplexiert zweiwertige Ionen, die für die Aktivität vieler Nukleasen notwendig sind. EDTA verzögert also den unerwünschten Abbau der Plasmid-DNA durch Nukleasen.
- Lagern Sie die isolierte Plasmid-DNA immer bei mindestens -20°C im Kühlschrank. Portionieren Sie gegebenenfalls die plasmidhaltige Lösung, um nicht ständig auf eine einzige Stammlösung zugreifen und sie auftauen und frieren zu müssen.
- Verwenden Sie immer sterile Pipettenspitzen, um Teilvolumina einer Plasmid-Stammlösung zu entnehmen. Überall, wo lebensfähige Bakterien vorkommen, ist auch mit größeren Mengen Nukleasen zu rechnen, die die Plasmid-DNA unkontrolliert abbauen.
Unterlagen
Die Zahlen geben die Dateigrößen in KB wieder. Zum Lesen der pdf-Dateien benötigen Sie den Adobe Acrobat Reader (einige Dateien wurden für Version 7 oder besser optimiert!). Zum Betrachten der ppt-Dateien benötigen Sie Microsofts PowerPoint. Deren Ausdruck als pdf-Datei erlaubt Ihnen die Herstellung von Folien für den Tageslichtprojektor.
Tipp: Laminieren Sie solche Seiten, die Sie häufiger als Arbeitsunterlagen nutzen möchten.
| Folgeexperimente | 35 KB | |
| Isolierung + Erklärungen | 37 KB | |
| Methode | 440 KB | 2400 KB |
| Übersicht | 38 KB | |
| Verfahren | 38 KB | |
| pUC18 Genkarte | 30 KB | |
| pUC18 Nukleotidsequenz | 170 KB | |
| pBR322 Genkarte | 38 KB | |
| pBR322 Nukleotidsequenz | 170 KB | |
| pBluescript II KS+ Genkarte | 54 KB |
Fragen
- Wie kann prinzipiell eine Nukleotidsequenz so abgeändert werden, dass die kodierten Aminosäuren zwar erhalten bleiben, an der geänderten Nukleotidsequenz jedoch eine Schnittstelle für eine Restriktionsendonuklease entsteht?
- Wie groß ist der Anteil von 500 Kopien pUC18-Nukleinsäuremolekülen (3 kBp = 3x103 Bp pro Molekül) an der gesamten chromosomalen DNA (4,5 x 106 Bp pro Genomkopie) einer E. coli Zelle?
- Wie hoch ist der prozentuale Anteil des gesamten Zellproteins und der chromosomalen DNA am Trockengewicht einer E. coli Zelle?
- Warum kann ein Plasmid in größerer Kopienzahl pro Zelle vorkommen, als das Genom?
- Kann jedes Plasmid in jeder Bakterienart vermehrt werden?
