Polymerasekettenreaktion (PCR)
Ergebnisse
Da die PCR-Amplifikate des 16S rRNA Gens aus sehr vielen Bakterien etwa 1450 Bp lang sind, kann die einfache Fragmentlängenbestimmung in der Agarosegelelektrophorese nicht für eine Unterscheidung der Organismen verwendet werden. Eine eindeutige Unterscheidung wird erst möglich durch die Nukleotidsequenzierung. Da dieser Ansatz für Reihenuntersuchungen (z.B. Isolierung neuer Stämme) teuer und aufwendig ist, wird ersatzweise das Muster ("fingerprint") nach Verdau mit Restrikionsendonukleasen ausgewertet. Die Längen aller Teilfragmente aus dem Verdau addieren sich auf ca. 1450 Bp. Wird die Summe größer, so handelt es sich um eine Mischkultur, die also mindestens zwei Amplifikatspezies in einem PCR-Ansatz lieferte.
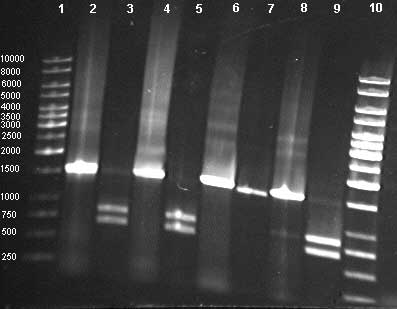
Abbildung 1: Elektrophoretische Analyse von PCR-Produkten in einem 0,8% Agarosegel, gefärbt mit Ethidiumbromid. Spuren 1 und 10: Längenstandard ; Spuren 2,4,6,8 : unverdaute PCR-Produkte; Spuren 3,5,7 und 9 mit Eco RI verdaute Produkte.
Dieses experimentell gefundene Muster stimmt mit dem theoretischen Muster überein, das durch den NEBcutter für den Verdau des 16S rRNA Genamplifikates von Bacillus subtilis mit Eco RI vorausgesagt wird. Die Nukleotidsequenz dieses Gens ist im Abschnitt "Nucleotide" auf dem NCBI-Server unter der Nummer AY134870 zu finden. Nach Eco RI-Verdau sollten zwei Fragmente mit den Längen 859 und 582 Bp entstehen.
Die entsprechende Nukleotidsequenz für das Gen aus E. coli ist dort unter der Nummer J01859 zu finden.