Kurzfassung zur Methode
- PCR-Ansatz mit Ready-to-go beads:
- 18 µl steriles Wasser pro Gefäß
- jeweils 1 µl Primer
- 5 µl template-DNA (eventuell verdünnte chromosomale DNA)
- Gesamtvolumen: 25 µl pro Reaktionsgefäß
Programmierung des PCR-Gerätes:
- 3 min bei 95°C (Denaturierung der DNA)
- dann 35 Zyklen mit:
- 90 sec bei 94°C (schmelzen der DNA-Doppelstränge)
- 90 sec bei 50°C (oder die empfohlene Hybridisierungstemperatur für ihr Primerpaar)
- 2 min bei 72°C (Polymerisationsreaktion)
- abschließend 30 min bei 72°C (komplettieren der Polymerisationsreaktion)
- eventuell gefolgt von einem Kühlschritt bei 4°C, falls der Ansatz nicht sofort weiterverarbeitet und z.B. über Nacht inkubiert wird.
Hinweise für die Entwicklung einer eigenen PCR
Sie könnten wie folgt vorgehen:
- Sie wählen aus einer Datenbank (z.B. ncbi) die DNA-Sequenz eines Gens.
- Sie geben diese Nukleotidsequenz in das im Internet verfügbar Programm Primo ein.
- Lassen Sie die dort vorgesehenen Optionen zunächst unverändert.
- Nach wenigen Sekunden bekommen Sie als Ergebnis die Nukleotidsequenz des "forward" und des "reverse" Primers sowie die zu erwartende Länge des PCR-Ampifikates mitgeteilt.
- Geben Sie diese Nukleotidsequenzen bei Ihrer Synthesefirma (z.B. Biomers, MWG) in Auftrag.
- Testen Sie Ihre primer in der PCR: Die Synthesefirma gibt ihnen wie das Internet-Programm Hinweise auf die Annealing-Temperatur (notfalls beginnen Sie mit etwa 5°C niedrigerer Temperatur, als vorausberechnet wurde.)
Fehlerraten von DNA-Polymerasen
DNA-Polymerasen arbeiten nicht fehlerfrei. Je nach Verwendung des PCR-Produktes (Amplifikates) ist jedoch eine geringe Fehlerrate erforderlich. Eine englischsprachige Veröffentlichung zu diesem Thema gibt es hier.
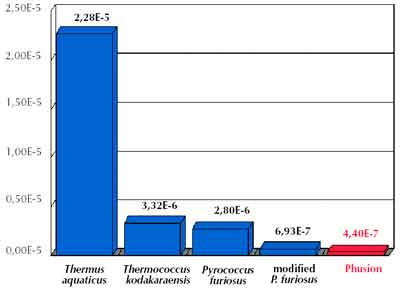
Abbildung 1: Fehlerraten verschiedener DNA-Polymerasen. Es ist erkennbar, dass die Taq-Polymerase aus Thermus die häufigtsen, die Phusion-Polymerase die wenigsten Fehler liefert.
Fehl-Amplifikationen werden auch durch einen so genannten hot-start vermieden:
Berechnung zur Amplifikation in der PCR
Die PCR ist ein Prozess, bei dem eine lineare sowie eine exponenzielle Vermehrung einer Vorlagen-DNA (template) stattfindet. Die exponenzielle Zunahme liefert das eigentliche "PCR-Produkt". Der Anteil aus der linearen Vermehrung kann in der Regel nicht detektiert werden, weil für die gebräuchlichen Nachweismethoden zu wenige Moleküle, und die auch noch in variablen Längen gebildet werden.
Da es sich bei der exponenziellen Vermehrung um eine Verdopplung in jedem Syntheseschritt handelt, kann die Anzahl der gebildeten Moleküle wie das Wachstum einer Bakterienkultur durch eine geometrische Progession berechnet werden. Aus 1 werden 2 (21), daraus 4 (22), dann 8 (23) Kopien (Bakterien) gebildet, oder allgemein 2n. Wenn wir für n = 30 Synthesezyklen ansetzen, dann werden also 230 = 1,07 x 109 Kopien aus einer Vorlage gebildet. Bei 35 Zyklen sind es bereits 3,4 x 1010 Kopien.
In der Praxis ist jedoch die Amplifikation nur eines template-Moleküls graue Theorie. Gestartet wird mit mehreren (> 100) Molekülen.
Klonierung von PCR-Produkten
Manchmal ist es erforderlich, die PCR-Produkte z.B. auf Ihre Nukleotidsequenz hin zu untersuchen. Dazu gibt es zwei Möglichkeiten:
- Die PCR-Produkte werden gereinigt und sofort sequenziert. (Funktioniert in der Praxis leider nicht immer).
- Die PCR-Produkte werden in einen Vector kloniert und dann sequenziert. Dazu werden die Primer mit flankierenden Nukleotidsequenzen versehen, die RE-Schnittstellen am 5`-Ende enthalten. Nach der Amplifikation wird das PCR-Produkt mit der entsprechenden Restriktionsendonuklease behandelt und diese Produkte in einen Klonierungsvector ligiert, dessen multiple Klonierungsstelle ebenfalls mit dieser Restriktionsendonuklease geöffnet wurde.
Gerichtet Mutagenese durch PCR
Durch die PCR kann auch eine gerichtet Mutagenese (site-directed mtagenesis) durchgeführt werden. Dazu gibt es einen englischsprachigen Artikel.
Nested PCR
Bei sehr geringen Mengen Template (DNA, die als Vorlage für die PCR dient) wird eine verschachtelte PCR durchgeführt. Auf eine "normale" PCR wird eine weitere mit weiter innen liegenden Primern aufgesetzt.
Herstellen von cDNA
Die Expression eines Genes kann auch über eine RT-PCR (PCR mit Reverser Transkriptase) durchgeführt werden. Dazu wird die mRNA in einem ersten Schritt durch das virale Enzym Reverse Transkriptase (aus RNA-Viren!) in cDNA (copy DNA) umgeschrieben, mit der dann eine "normale PCR" durchgeführt wird. Bei der vorgeschalteten mRNA Isolierung wird natürlich die vorhandene chromosomale DNA abgetrennt.
Hot-start PCR
Heute werden überwiegend rekombinante DNA-Polymerasen eingesetzt. Die Veränderungen am Enzym gehen so weit, dass deren Aktivität bei niederigen Temperaturen durch einen Antikörper oder ein anderes Protein gehemmt ist. Erst bei hohen Temeperaturen wird diese Hemmung aufgehoben. Der Vorteil: alle DNA-Polymerasen beginnen in einen engen, hohen Temperaturbereich zu arbeiten, was zu weniger Fehlbindungen der Primer (bei niedrigen Temperaturen) führt.
